I complessi multienzimatici sono strutture discrete e stabili formate da enzimi associati in modo non covalente che catalizzano due o più reazioni consecutive di una via metabolica.[7]
Possono essere considerati come un passo in avanti nell’evoluzione dell’efficienza catalitica in quanto assicurano vantaggi che i singoli enzimi, anche quelli che hanno raggiunto la perfezione catalitica, da soli non avrebbero.[1][12]
Indice
Quali vantaggi offrono i complessi multienzimatici?
Nel corso dell’evoluzione alcuni enzimi si sono evoluti sino al raggiungimento della perfezione catalitica. Si tratta cioè di enzimi per i quali quasi ogni collisione con il proprio substrato ne determina la conversione in prodotto. Esempi sono:
- la fumarasi (EC 4.2.1.2), che catalizza la settima tappa del ciclo dell’acido citrico, ossia l’idratazione/deidratazione reversibile del doppio legame del fumarato a dare malato;
- l’acetilcolinesterasi (EC 3.1.1.7), che catalizza l’idrolisi dell’acetilcolina, un neurotrasmettitore, in colina e acido acetico, che a sua volta si dissocia a dare acetato e ioni idrogeno;
- la superossido dismutasi (EC 1.15.1.1), che catalizza la conversione, e quindi l’inattivazione, del radicale superossido (O2.-), una specie molto reattiva, in perossido d’idrogeno od acqua ossigenata (H2O2) e acqua;
- la catalasi (EC 1.11.1.6), che catalizza la degradazione di H2O2 in acqua e ossigeno.
La velocità con cui avviene una reazione enzimatica è dunque in parte è determinata dalla frequenza con cui i substrati e gli enzimi stessi collidono. Ne deriva che un modo semplice per aumentarla sia quello di aumentare le concentrazioni di enzima e substrato. Tuttavia, visto l’enorme numero di reazioni che si verificano all’interno della cellula le loro concentrazioni non potranno essere elevate. E infatti nella cellule la concentrazione della maggior parte dei metaboliti è dell’ordine delle micromoli (10-6 M), e per la maggior parte degli enzimi anche più bassa.[1]
L’evoluzione ha quindi imboccato strade differenti per aumentare la velocità di reazione. Una delle quali è stata quella di ottimizzare l’organizzazione spaziale degli enzimi con la formazione dei complessi multienzimatici e degli enzimi multifunzionali, ossia strutture che permettono di minimizzare la distanza che il prodotto di una reazione deve percorrere per passare al sito attivo che catalizza la reazione successiva, essendo i siti attivi vicini gli uni agli altri.[2][7]Si verifica cioè l’incanalamento dei substrati, in inglese substrate o metabolic channeling,[8][9][14] incanalamento che può avvenire anche attraverso veri e propri canali intramolecolari che connettono i siti attivi, come nel caso, tra i complessi enzimatici, del complesso della triptofano sintasi (EC 4.2.1.20),[3][4]il cui tunnel fu il primo a essere scoperto, e quello della carbamil fosfato sintetasi batterica (EC 6.3.4.16).[5][13]
L’incanalamento dei substrati è in grado di aumentare la velocità di reazione, ma più in generale l’efficienza catalitica, in più modi, di seguito brevemente descritti.[8][12]</sup>
- Viene limitata al minimo la diffusione di substrati e prodotti nel mezzo circostante, quindi la loro diluizione e riduzione della concentrazione, producendo anzi elevate concentrazioni locali anche quando la loro concentrazione cellulare è bassa, il che porta a un aumento della frequenza delle collisioni enzima-substrato.
- Viene ridotto il tempo di transito dei substrati da un sito attivo al sito attivo successivo.
- Viene ridotta la probabilità che si verifichino reazioni collaterali.
- Gli intermedi chimicamente labili sono protetti dalla degradazione da parte del solvente.
Un altro vantaggio metabolico apportato dai complessi multienzimatici, analogamente a quanto accade con gli enzimi multifunzionali, è che consentono di controllare in modo coordinato l’attività catalitica degli enzimi che lo compongono. E se si aggiunge il fatto che spesso l’enzima che catalizza la prima reazione della sequenza è l’enzima regolatorio, è possibile evitare:
- la sintesi di intermedi non necessari, altrimenti prodotti se la sequenza di reazioni fosse regolata a valle della prima reazione;
- la sottrazione di metaboliti ad altre vie come pure uno spreco di energia.
Esempi
Da quanto detto in precedenza non sorprende che, specialmente nelle cellule eucariote, i complessi multienzimatici, al pari degli enzimi multifunzionali, siamo comuni e coinvolti in differenti vie metaboliche, sia anaboliche che cataboliche, mentre sono pochi gli enzimi liberamente diffusibili. Di seguito alcuni esempi.
Alfa-chetoacido deidrogenasi
Esempi classici di complessi multienzimatici sono i tre complessi appartenenti alla famiglia delle alfa-chetoacido deidrogenasi o 2-ossiacido deidrogenasi, ossia:
- il complesso della piruvato deidrogenasi o PDC, acronimo dell’inglese pyruvate dehydrogenase complex;[2]
- il complesso dell’α-chetoacido deidrogenasi a catena ramificata o BCKDH , acronimo dell’inglese branched-chain α-keto acid dehydrogenase complex;
- il complesso della α-chetoglutarato deidrogenasi, o 2-oxoglutarato deidrogenasi o OGDH, acronimo dell’inglese 2-oxoglutarate dehydrogenase.[2][6][7][10]
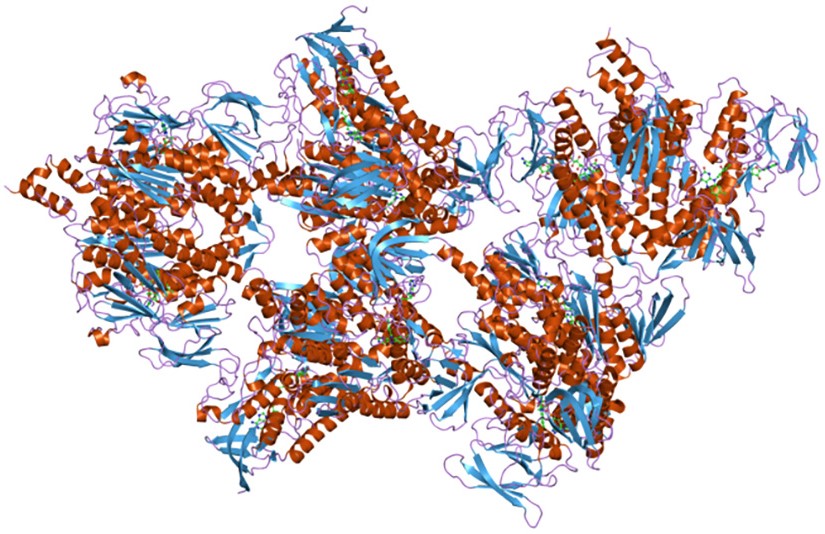
I tre complessi sono correlati sia dal punto di vista strutturale che funzionale.
Considerando ad esempio il complesso della piruvato deidrogenasi questi è formato da copie multiple di tre enzimi differenti:
- piruvato deidrogenasi o E1 (EC 1.2.4.1);
- diidrolipoil transacetilasi o E2 (EC 2.3.1.12);
- diidrolipoil deidrogenasi o E3 (EC 1.8.1.4).
PDC, sia nei procarioti che negli eucarioti presenta quindi una struttura di base E1-E2-E3, struttura che si ritrova anche negli altri due complessi.[15] In aggiunta, all’interno di una data specie:
- la diidrolipoil deidrogenasi è identica;
- la piruvato deidrogenasi e la diidrolipoil transacetilasi sono omologhe
E, sebbene questi enzimi siamo specifici per i rispettivi substrati, utilizzano gli stessi cofattori, ossia il coenzima A, il NAD, al tiamina pirofosfato, il FAD e la lipoamide.
Al fine di differenziarle vengono indicate, per il complesso della piruvato deidrogenasi, della α-chetoglutarato deidrogenasi e della α-chetoacido deidrogenasi a catena ramificata, rispettivamente come:
- E1p, E1o ed E1b (EC 1.2.4.4);
- E3p, E3o, and E3b (EC 1.8.1.4).</li>
Nota: il complesso della piruvato deidrogenasi degli eucarioti è il più grande complesso multienzimatico conosciuto, più grande di un ribosoma e visibile anche al microscopio elettronico.[2]
Il complesso della piruvato deidrogenasi, che rappresenta il ponte di collegamento tra glicolisi e ciclo dell’acido citrico, catalizza la decarbossilazione ossidativa del piruvato, che è la base coniugata dell’acido piruvico e fa parte del gruppo dei chetoacidi, in particolare è un alpha-chetoacido. Nel corso delle reazioni il gruppo carbossilico del piruvato viene rilasciato in forma di anidride carbonica (CO2) e si verifica il trasferimento del gruppo acetilico risultante al coenzima A a dare acetil-coenzima A. Inoltre sono rilasciati due elettroni che sono trasferiti al NAD+.
Anche nel corso delle reazioni catalizzate dal complesso della α-chetoglutarato deidrogenasi e dal complesso della α-chetoacido deidrogenasi a catena ramificata, rispettivamente quarta reazione del ciclo dell’acido citrico, ossia l’ossidazione dell’α-chetoglutarato a succinil-CoA, e l’ossidazione degli α-chetoacidi derivanti dal catabolismo degli amminoacidi a catena ramificata valina, leucina e isoleucina, si verifica:
- la liberazione del carbonio carbossilico dell’alfa-chetoacido in forma di CO2;
- il trasferimento del gruppo acilico risultante al coenzima A a dare l’acil-CoA corrispondente;
- la riduzione del NAD+ a NADH.
Dunque, la notevole somiglianza esistente tra le strutture proteiche, i cofattori richiesti e i meccanismi di reazione riflettono senza dubbio una origine evolutiva comune.
Triptofano sintasi
Il complesso della triptofano sintasi è uno degli esempi di incanalamento dei substrati meglio studiati.[3][12][14] Presente nei batteri e nelle piante ma non negli animali, nei batteri è formato da due subunità α e due β assemblate in unità dimeriche αβ, che sembrano esser l’unità funzionale del complesso, a dare una struttura tetramerica αββα.[4][5][8]
Il complesso catalizza gli ultimi due passaggi della sintesi del triptofano: dall’indolo-3-glicero fosfato, per azione di una liasi (EC 4.1.2.8) presente sulle subunità α, viene liberata una molecola di gliceraldeide-3-fosfato e l’indolo. L’indolo diffonde quindi verso il sito attivo presente sulla subunità β sfruttando un tunnel idrofobico di circa 30 Å che, in ciascuna coppia αβ, connette i due siti attivi. Nel sito attivo della subunità β avviene, in presenza del piridossal-5-fosfato, la condensazione tra l’indolo e una serina a dare il triptofano.
Acetil-CoA carbossilasi
L’acetil-CoA carbossilasi o ACC (EC 6.4.1.2), membro della famiglia delle carbossilasi biotina-dipendenti, catalizza la prima tappa di comando della sintesi degli acidi grassi, ossia la carbossilazione dell’acetil-CoA a malonil-CoA, che è utilizzato come donatore di unità bicarboniose dalla acido grasso sintasi (EC 2.3.1.85) durante il processo di allungamento che porterà alla sintesi dell’acido palmitico.[9][11][12]
Nei batteri ACC è un complesso multienzimatico composto da due enzimi, la biotina carbossilasi (EC 6.3.4.14) e una carbossitransferasi, cui si aggiunge una proteina trasportatrice della biotina o BCCP, acronimo dell’inglese biotin carboxyl-carrier protein.
Nei mammiferi e negli uccelli è invece un enzima multifunzionale, in quanto le due attività enzimatiche sono presenti su una stessa catena polipeptidica, nella quale si ritrova anche BCCP.[2]
Nelle piante superiori sono presenti entrambe le forme.
Carbamil fosfato sintetasi
Altro esempio ben caratterizzato di incanalamento dei substrati è il complesso della carbamil fosfato sintetasi dei batteri,[9][12][14] che catalizza la sintesi del carbamil fosfato, necessario sia per la sintesi delle pirimidine che per quella dell’arginina. Il complesso presenta un tunnel lungo circa 100 Å all’interno del quale si muovono i prodotti delle sue tre attività enzimatiche.[13]
La prima catalizza la cessione dell’azoto ammidico della glutammina in forma di ione ammonio, il quale entra nel tunnel dove nel secondo sito attivo si combina con il bicarbonato, a spese di una molecola di ATP, a dare carbamato, che infine nell’ultimo sito attivo viene fosforilato a carbamil fosfato.
Bibliografia
- ^ a b Alberts B., Johnson A., Lewis J., Morgan D., Raff M., Roberts K., Walter P. Molecular biology of the cell. 6th Edition. Garland Science, Taylor & Francis Group, 2015
- ^ a b c d e Garrett R.H., Grisham C.M. Biochemistry. 4th Edition. Brooks/Cole, Cengage Learning, 2010
- ^ a b Hilario E., Caulkins B.G., Huang Y-M. M., You W., Chang C-E. A., Mueller L.J., Dunn M.F., and Fan L. Visualizing the tunnel in tryptophan synthase with crystallography: insights into a selective filter for accommodating indole and rejecting water. Biochim Biophys Acta 2016;1864(3):268-279. doi:10.1016/j.bbapap.2015.12.006
- ^ a b Hyde C.C., Ahmed S.A., Padlan E.A., Miles E.W., and Davies D.R. Three-dimensional structure of the tryptophan synthase multienzyme complex from Salmonella typhimurium. J Biol Chem 1988;263(33):17857-17871. doi:10.1016/S0021-9258(19)77913-7
- ^ a b Hyde C.C., Miles E.W. The tryptophan synthase multienzyme complex: exploring structure-function relationships with X-ray crystallography and mutagenesis. Nat Biotechnol 1990:8;27-32. doi:10.1038/nbt0190-27
- ^ Koolman J., Roehm K-H. Color atlas of Biochemistry. 2nd Edition. Thieme, 2005
- ^ a b c Michal G., Schomburg D. Biochemical pathways. An atlas of biochemistry and molecular biology. 2nd Edition. John Wiley J. & Sons, Inc. 2012
- ^ a b c Moran L.A., Horton H.R., Scrimgeour K.G., Perry M.D. Principles of Biochemistry. 5th Edition. Pearson, 2012
- ^ a b c Nelson D.L., Cox M.M. Lehninger. Principles of biochemistry. 6th Edition. W.H. Freeman and Company, 2012
- ^ ^ Perham R.N., Jones D.D., Chauhan H.J., Howard MJ. Substrate channeling in 2-oxo acid dehydrogenase multienzyme complexes. Biochem Soc Trans 2002;30(2):47-51. doi:10.1042/bst0300047
- ^ Rodwell V.W., Bender D.A., Botham K.M., Kennelly P.J., Weil P.A. Harper’s illustrated biochemistry. 30th Edition. McGraw-Hill Education, 2015
- ^ a b c d e Voet D. and Voet J.D. Biochemistry. 4th Edition. John Wiley J. & Sons, Inc. 2011
- ^ a b Yon-Kahn J., Hervé G. Molecular and cellular enzymology. Springer, 2009
- ^ a b c Welch G. R., Easterby J.S. Metabolic channeling versus free diffusion: transition-time analysis. Trends Biochem Sci 1994;19(5):193-197. doi:10.1016/0968-0004(94)90019-1
- ^ Zhou Z.H., McCarthy D.B., O’Connor C.M., Reed L.J., and J.K. Stoops. The remarkable structural and functional organization of the eukaryotic pyruvate dehydrogenase complexes. Proc Natl Acad Sci USA 2001;98(26):14802-14807. doi:10.1073/pnas.011597698