Il tratto gastrointestinale dell’uomo è una delle più ostili e competitive nicchie ecologiche presenti in natura. Ospita un ecosistema complesso, composto da virus, eucarioti, batteri e membri degli Archaea, incluso Methanobrevibacter smithii. Questo ecosistema, collettivamente denominato microbiota intestinale, rappresenta una componente importante del più ampio microbiota umano.[1][2]
Il microbiota intestinale è caratterizzato da una notevole complessità tassonomica e funzionale. Comprende centinaia di specie microbiche organizzate in comunità strutturate, plasmate dalle interazioni ecologiche tra microrganismi, inclusi batteri, batteriofagi e altri componenti dell’ecosistema intestinale. Insieme, queste interazioni contribuiscono sia alla stabilità della comunità che alla variabilità interindividuale.[3][4]
Oltre alle caratteristiche riguardanti la sua composizione, il microbiota intestinale è un’entità altamente dinamica rispetto alla variabile temporale. Le comunità microbiche si stabiliscono precocemente nel corso della vita, subiscono un rapido rimodellamento durante l’infanzia e la fanciullezza e si evolvono progressivamente durante l’età adulta e l’invecchiamento. Gli eventi della prima infanzia, in particolare la modalità di parto, svolgono un ruolo fondamentale nel suo assemblaggio iniziale e possono influenzare lo sviluppo del sistema immunitario e le traiettorie microbiche a lungo termine.[5][6][7]
Dopo questa fase di sviluppo precoce, ulteriori fattori influenzano progressivamente la composizione e la funzione del microbiota intestinale. Tra questi, le differenze spaziali lungo il tratto gastrointestinale, determinate da variazioni di pH, di disponibilità di ossigeno, dagli enzimi digestivi e sali biliari, nonché da agenti esterni come la dieta e la posizione geografica. Insieme, queste pressioni selettive contribuiscono alla diversificazione delle comunità microbiche e alla loro specializzazione funzionale.[8][9][10]
Di conseguenza, il microbiota intestinale emerge come un ecosistema dinamico, organizzato spazialmente e regolato temporalmente. In quanto componente centrale del microbiota umano, svolge un ruolo fondamentale nella fisiologia dell’ospite e rappresenta un collegamento chiave tra esposizione ambientale, biologia dell’ospite e salute e malattia.[11]
Indice
- Composizione del microbiota intestinale
- Composizione del microbiota intestinale nel corso della vita
- Microbiota intestinale e ambiente intestinale
- Microbiota intestinale e dieta
- Microbiota intestinale, stato di salute e malattie
- Microbiota intestinale, batteriocine e batteriofagi
- Microbiota intestinale e posizione geografica
- Bibliografia
Composizione del microbiota intestinale
L’abbondanza e la composizione batterica variano lungo il tratto gastrointestinale e risultano particolarmente elevate nel colon.
Il colon umano adulto ospita più di 400 specie batteriche, appartenenti a nove phylum principali dei circa 30 attualmente riconosciuti. Tuttavia, approcci metagenomici suggeriscono che la diversità reale sia sostanzialmente più elevata.[12]
Nella tabella seguente sono riassunti i phylum batterici più abbondanti del microbiota intestinale umano adulto occidentale e i loro generi rappresentativi.[13][14]
| Phylum | Colorazione di Gram | Generi rappresentativi |
|---|---|---|
| Actinobacteria | Gram-positivi | Bifidobacterium, Collinsella, Corynebacterium, Eggerthella, Propionibacterium |
| Bacteroidetes | Gram-negativi | Più di 20 generi, inclusi Bacteroides e Prevotella |
| Cyanobacteria | Gram-negativi | — |
| Firmicutes | Gram-positivi | Mycoplasma, Bacillus, Clostridium, Dorea, Faecalibacterium, Ruminococcus, Eubacterium, Staphylococcus, Streptococcus, Lactobacillus, Lactococcus, Enterococcus, Sporobacter, Roseburia |
| Fusobacteria | Gram-negativi | — |
| Lentisphaerae | Gram-negativi | — |
| Proteobacteria | Gram-negativi | Escherichia, Klebsiella, Shigella, Salmonella, Citrobacter, Helicobacter, Serratia |
| Spirochaetes | Gram-negativi | — |
| Verrucomicrobia | Gram-negativi | Akkermansia |
Ecosistema intestinale e viroma
L’intestino degli adulti ospita un’ampia e varia comunità di virus a DNA e RNA, collettivamente nota come viroma, comprendente migliaia di genotipi virali, nessuno dei quali è dominante. Infatti, il virus più abbondante rappresenta solo circa il 6% della comunità, al contrario di ciò che accade nei neonati dove il genotipo più abbondante ne rappresenta oltre il 40%.[15][16][17]
La maggior parte dei virus a DNA sono batteriofagi o fagi, ossia virus che infettano i batteri. I fagi sono anche l’entità biologica più abbondante presente sulla terra, con una popolazione stimata di circa 1031 unità, mentre la maggior parte di quelli a RNA sono virus vegetali.[18]
Pressione selettiva e variabilità individuale
La presenza nel colon di solo un piccolo sottoinsieme del mondo batterico è il risultato di forti pressioni selettive che hanno agito durante l’evoluzione sia sui colonizzatori microbici, selezionando organismi altamente adattati a questo ambiente, sia sulla nicchia intestinale stessa.[3][7] Tuttavia, ogni individuo ospita nell’intestino una comunità batterica unica. Questa specificità individuale coesiste con modelli ecologici ricorrenti osservati nelle popolazioni.[19]
Nonostante la grande variabilità esistente sia riguardo ai taxa presenti che tra gli individui, è stato proposto, ma non da tutti accettato, che nella maggior parte degli adulti il microbiota intestinale, nella sua componente batterica, possa essere classificato in varianti o enterotipi, in base all’abbondanza relativa dei generi Bacteroides e Prevotella.[20] Questo suggerisce che esista un numero limitato di stati simbiotici ben bilanciati, che potrebbe rispondere in maniera differente a fattori quali la dieta, l’età, la genetica, lo stato di salute e l’assunzione di farmaci.[21]
Composizione del microbiota intestinale nel corso della vita
Lo sviluppo dell’ecosistema microbico intestinale è un evento complesso e cruciale nella vita dell’uomo e altamente variabile da individuo a individuo.[6][7]
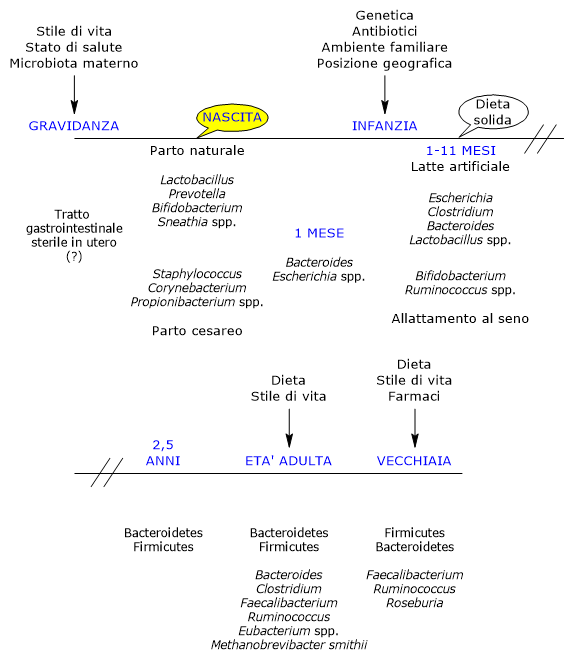
Nell’utero, l’intestino è stato tradizionalmente considerato sterile; tuttavia, viene rapidamente colonizzato dai microbi alla nascita, poiché il neonato nasce con una tolleranza immunologica modellata dalla madre.[22]
Tuttavia, alcuni lavori stanno evidenziando la presenza di batteri nel tessuto placentare, nel sangue del cordone ombelicale, nelle membrane fetali e nel liquido amniotico di neonati sani senza segni di infezioni o infiammazioni.[23] Ad esempio, il meconio di neonati prematuri nati da madri sane contiene un microbiota specifico, con i Firmicutes come phylum principale e predominanza di stafilococchi. Di contro, nelle prime feci i più abbondanti sono i Proteobacteria, in particolare specie quali Escherichia coli, Klebsiella pneumoniae, Serratia marcescens, ma anche gli enterococchi.[24]
Sembra che sia i batteri vaginali che quelli intestinali possano avere accesso al feto, anche se attraverso vie differenti: per via ascendente i primi, tramite le cellule dendritiche del sistema immunitario i secondi. Dunque, è stata proposta anche l’esistenza di un microbiota fetale.[6]
Modalità del parto
Il primo determinante fondamentale della colonizzazione microbica postnatale è la modalità del parto.[25]
Alla nascita si verifica la colonizzazione dell’intestino neonatale ad opera di un piccolo inoculo di origine materna, costituito prevalentemente da batteri aerobi obbligati e facoltativi, poiché inizialmente l’intestino del neonato contiene ossigeno. Questi sono poi sostituiti da anaerobi obbligati, tipicamente presenti nell’età adulta, cui i primi colonizzatori hanno preparato l’ambiente.[26]
Nei bambini nati da parto naturale, le principali fonti di inoculo sono il microbiota vaginale e fecale materno. I neonati ospitano infatti comunità microbiche dominate da specie dei generi Lactobacillus, particolarmente abbondanti nel microbiota vaginale e nell’intestino neonatale nei primi giorni, Bifidobacterium, Prevotella e Sneathia.[27] Gli anaerobi, come i membri dei Bacteroidetes e Firmicutes, non potendo vivere all’esterno del loro ospite, dipendono probabilmente dal contatto ravvicinato madre–figlio per la trasmissione. Inoltre, a causa della presenza di ossigeno nell’intestino del neonato, la trasmissione degli anaerobi stretti potrebbe non avvenire alla nascita, ma in seguito, verosimilmente tramite spore.[28]
In caso di taglio cesareo, i primi batteri incontrati dal neonato sono prevalentemente quelli della cute e dell’ambiente ospedaliero. Il microbiota intestinale è dominato da specie appartenenti ai generi Corynebacterium, Staphylococcus e Propionibacterium, con una minore carica batterica complessiva e una ridotta diversità nelle prime settimane di vita rispetto ai neonati nati da parto naturale.[29][30]
Un’ulteriore evidenza a sostegno della trasmissione verticale è rappresentata dalla somiglianza tra il microbiota del meconio e quello dei potenziali siti materni. Tali “firme” microbiche non persistono indefinitamente, ma vengono sostituite entro il primo anno di vita.[31]
Fonti successive di inoculo includono la bocca e la pelle dei familiari, oggetti e animali; tuttavia, il latte materno sembra svolgere un ruolo centrale nel determinare la successione microbica intestinale.
La variabilità osservata tra i bambini rifletterebbe l’individualità di queste esposizioni.[32]
Modalità del parto e sistema immunitario
Il tipo di parto sembra influenzare anche lo sviluppo del sistema immunitario nel corso del primo anno di vita, forse attraverso i suoi effetti sul microbiota intestinale.[33]
I bambini nati con il taglio cesareo mostrano:
- una più bassa conta di cellule batteriche nei campioni di feci a un mese dalla nascita, dovuta soprattutto alla ridotta presenza di Bifidobacterium;
- un più alto numero di cellule secernenti anticorpi, il che potrebbe riflettere un’eccessiva esposizione agli antigeni a seguito di un’aumentata permeabilità intestinale.
Nel loro insieme, questi risultati supportano il concetto che le alterazioni precoci della colonizzazione microbica associate al parto cesareo possano influenzare la maturazione del sistema immunitario durante una finestra critica dello sviluppo.[30][34][35]
Dalla nascita al primo mese
Entro pochi giorni dalla nascita si viene a creare una fiorente comunità la cui popolazione è molto più variabile in composizione e meno stabile nel tempo rispetto a quella dell’adulto, e che ben presto supererà in numero quella delle cellule del neonato stesso, evolvendo secondo un pattern temporale notevolmente variabile da individuo a individuo.[5]
Durante il primo mese di vita è presente un limitato numero di taxa, principalmente appartenenti ai phylum Proteobacteria e Actinobacteria, che rimane relativamente stabile. Nei mesi successivi, tuttavia, si verifica un marcato aumento della variabilità, parallelo a quello di nuove varianti genetiche.[6] Numerosi studi sottolineano che l’esposizione iniziale ai microbi gioca un ruolo fondamentale nel definire le “traiettorie” che portano all’ecosistema adulto. Infine, queste comunità iniziali possono anche fungere da fonte diretta di batteri protettivi o patogeni.[17]
I virus, assenti alla nascita, dalla fine della prima settimana di vita raggiungono un numero di circa 108 unità per grammo di peso fresco di feci, e quindi rappresentano una componente abbondante e dinamica del microbiota intestinale in via di sviluppo. Sebbene la diversità virale sia inizialmente bassa, il viroma è dominato dai batteriofagi, che probabilmente influenzano l’abbondanza e la diversità batterica. L’origine di questi virus non è chiara; tra le possibili fonti ci sono ovviamente quelle materne e ambientali.[36] In particolare, i primi virus potrebbero essere il risultato dell’induzione dei profagi del microbiota intestinale del neonato, ipotesi avvalorata dall’osservazione che oltre il 25% delle sequenze fagiche sembrano essere molto simili a quelle dei fagi che infettano batteri come Lactobacillus, Lactococcus, Enterococcus e Streptococcus, generi abbondanti nel latte materno.[37][38]
Dal primo mese al primo anno
Dal termine del primo mese si ritiene che la fase iniziale di rapida acquisizione microbica seguente alla nascita sia terminata.
Nei bambini di un mese, i microrganismi più abbondanti appartengono ai generi Bacteroides ed Escherichia. Di seguito, i Bifidobacterium, assieme a Ruminococcus, aumentano fino a dominare l’intestino del bambino allattato al seno tra il primo e l’undicesimo mese di vita.[39][40]
I bifidobatteri, come Bifidobacterium longum subsp. infantis, sono:
- strettamente correlati all’allattamento al seno;
- tra i meglio caratterizzati fra i batteri commensali;
- probiotici, ossia microrganismi capaci di apportare effetti benefici alla salute dell’ospite.[41]
La loro abbondanza conferisce benefici anche attraverso un’esclusione competitiva con cui sono di ostacolo alla colonizzazione dei patogeni. Al contrario, batteri dei generi Escherichia, come E. coli, Clostridium, come C. difficile, Bacteroides, come B. fragilis e Lactobacillus sono presenti in modo significativamente maggiore nei bambini allattati con latte artificiale rispetto a quanto osservato nei bambini allattati al seno.[42]
Sebbene i neonati allattati al seno ricevano solo latte materno fino allo svezzamento, il loro microbiota può mostrare una notevole variabilità interindividuale sia nell’abbondanza tassonomica che nelle dinamiche temporali. Queste differenze potrebbero derivare da eventi casuali di colonizzazione, malattie, assunzione di antibiotici, cambiamenti nel comportamento dell’ospite o da altri aspetti legati allo stile di vita, come anche da differenze nelle risposte immunitarie ai microbi colonizzatori. Tuttavia, non è ancora chiaro come questi fattori contribuiscano a plasmare il microbiota intestinale del bambino.[5][43]
Sembra che anche il viroma si modifichi molto rapidamente dopo la nascita, poiché la maggior parte delle sequenze virali presenti nella prima settimana non si ritrova dopo la seconda, e la diversità si espande rapidamente durante i primi tre mesi.[17][44] Questo contrasta con la stabilità che si osserva nell’adulto, dove circa il 95% delle sequenze rimane stabile nel tempo.[15][45]
Dal primo anno ai due anni e mezzo
In condizioni normali, verso la fine del primo anno di vita, il neonato è stato esposto a una dieta complessa per un periodo significativo e ha sviluppato una comunità microbica con caratteristiche analoghe a quelle del microbiota intestinale adulto, quali:[5]
- una composizione più stabile, filogeneticamente più complessa, e progressivamente più simile tra soggetti diversi;[46]
- una preponderanza di Bacteroidetes e Firmicutes, seguiti dai Verrucomicrobia, e bassi livelli di Proteobacteria;[47]
- un aumento del carico batterico come dei livelli di acidi grassi a catena corta, principalmente l’acido acetico, l’acido propionico e l’acido butirrico, nelle feci;[48]
- un aumento dei geni associati al metabolismo dei carboidrati, alla biosintesi delle vitamine e alla degradazione degli xenobiotici.[49]
Interessante notare che il significativo turn-over di taxa che si verifica dalla nascita al termine del primo anno è accompagnato da una notevole costanza nelle capacità funzionali complessive.[50]
Verso la fine del primo anno di vita, anche i primi colonizzatori virali sono stati sostituiti da un viroma specifico del bambino.[44]
Verso i due anni e mezzo di vita il microbiota intestinale raggiunge la sua piena maturità, risultando molto simile a quello dell’adulto.
Questa maturazione è conseguenza di vari fattori, quali:
- il passaggio a una dieta per adulti;
- una maggiore idoneità all’ambiente intestinale dei taxa che tipicamente dominano il microbiota colonico adulto rispetto ai colonizzatori iniziali;
- i cambiamenti progressivi nell’ambiente intestinale, dovuti ai cambiamenti legati allo sviluppo della mucosa intestinale;
- gli effetti del microbiota stesso.
Di conseguenza, i primi 2–3 anni di vita sono il periodo più critico in cui intervenire al fine di ottenere il miglior microbiota possibile così da ottimizzare la crescita e lo sviluppo del bambino.[7]
Età adulta
Da uno stato inizialmente caotico, l’ecosistema intestinale si evolve in una configurazione adulta relativamente stabile, che persiste fino alla vecchiaia. Questo ecosistema include componenti virali, archeali ed eucariotiche e, nelle popolazioni occidentali, è dominato dai Firmicutes, che rappresentano circa il 60% della comunità batterica intestinale, seguiti dai Bacteroidetes e Actinobacteria, principalmente con il genere Bifidobacterium, ognuno con circa il 10% della comunità batterica, e quindi da Proteobacteria e Verrucomicrobia. I generi Bacteroides, Clostridium, Faecalibacterium, Ruminococcus ed Eubacterium costituiscono, assieme a Methanobrevibacter smithii, la grande maggioranza della comunità batterica intestinale dell’adulto.[51][52]
| Gruppo microbico | Phylum | Generi rappresentativi | Abbondanza approssimativa |
|---|---|---|---|
| Batteri | Firmicutes | Clostridium, Faecalibacterium, Ruminococcus, Eubacterium, Roseburia | ~60% |
| Batteri | Bacteroidetes | Bacteroides, Prevotella | ~20–30% |
| Batteri | Actinobacteria | Bifidobacterium, Collinsella | ~10% |
| Batteri | Proteobacteria | Escherichia, Klebsiella, Helicobacter | <5% |
| Batteri | Verrucomicrobia | Akkermansia | <5% |
| Archaea | Euryarchaeota | Methanobrevibacter | <1% |
| Virus | — | Batteriofagi | Molto abbondante |
Modelli differenti sono invece stati osservati in popolazioni rurali dell’Africa.[53] Tuttavia, il microbiota intestinale è risultato sufficientemente conservato tra gli individui da consentire l’identificazione di microbioma centrale condiviso.[54]
La stabilità e resilienza sono influenzate da numerose variabili, tra cui la dieta sembra essere una delle più importanti. Mantenere la stabilità del microbiota richiede quindi di ridurne al minimo i fattori di disturbo e prevenire le malattie, anche attraverso le vaccinazioni. Va comunque sottolineato che stabilità e resilienza potrebbero essere dannose nel caso in cui la comunità intestinale dominante sia patogena.[55][56]
Vecchiaia
Negli anziani, come già visto nei neonati, il microbiota intestinale va incontro a cambiamenti significativi.[57] Uno studio condotto in Irlanda su 161 persone sane di 65 o più anni, ha mostrato che, nella maggior parte dei soggetti, il microbiota intestinale differisce in modo sostanziale da quello degli adulti più giovani, con una composizione che è dominata dai Bacteroidetes, i principali, seguiti dai Firmicutes, con percentuali quasi inverse rispetto a quelle osservate negli adulti più giovani, sebbene siano state osservate notevoli variazioni tra i soggetti. Tra i generi più abbondanti si trovano Faecalibacterium, che rappresenta circa il 6% dei generi principali, seguiti dai Ruminococcus, Roseburia e Bifidobacterium, quest’ultimo intorno allo 0,4%.[58]
Anche la variabilità nella composizione delle comunità è più grande rispetto ai giovani adulti, il che potrebbe essere dovuto alla maggior morbilità associata con l’età, il maggior uso di farmaci, nonché a variazioni della dieta legate all’età.[51][59]
Microbiota intestinale e ambiente intestinale
Lo stomaco, a causa del pH estremamente acido del suo contenuto, è un ambiente ostile per i batteri, che sono presenti in numero relativamente basso, circa 102–103 cellule batteriche/grammo di tessuto. Oltre a Helicobacter pylori, in grado di causare gastriti e ulcere gastriche, sono presenti anche batteri appartenenti al genere Lactobacillus.[8][60]
Nel duodeno, si osserva un incremento nel numero di unità, circa 104–105 cellule batteriche/grammo di tessuto, e concentrazioni analoghe anche nel digiuno e nell’ileo prossimale.[61] Il numero contenuto di microorganismi presente nell’intestino tenue è dovuto all’ambiente inospitale conseguente al fatto che nel tratto discendente del duodeno è presente l’apertura dell’ampolla di Vater, dalla quale viene riversata la bile e il succo pancreatico, ossia sali biliari ed enzimi pancreatici, entrambi in grado di esercitare effetti antimicrobici che limitano la crescita batterica.[10]
Nell’ileo terminale, dove l’attività dei sali biliari e degli enzimi pancreatici è ridotta, la conta batterica è di circa 107 cellule batteriche/grammo di tessuto, fino ad arrivare nel colon a valori di 1012–1014 cellule batteriche/grammo di tessuto, tanto che le feci sono costituite per il 40% da batteri.[62]
Distribuzione batterica lungo l’intestino
L’ambiente intestinale, definito dai gradienti di pH, dai sali biliari, dagli enzimi digestivi, dalla disponibilità di ossigeno e dalla distribuzione dei nutrienti, esercita una notevole pressione selettiva sul microbiota intestinale.[63]
La distribuzione di batteri lungo l’intestino è strategica. Nel duodeno e nel digiuno, la quantità di nutrienti ancora disponibile è molto più alta rispetto a quella presente nell’ileo terminale e nel colon, dove sono rimasti acqua, fibre ed elettroliti. Dunque, non è un problema trovare nella parte distale dell’intestino elevate densità batteriche.[64] Al contrario, concentrazioni batteriche elevate nel duodeno, nel digiuno e nell’ileo prossimale sono patologiche e caratterizzano una condizione nota come sovracrescita batterica dell’intestino tenue (SIBO), in cui il numero di batteri aumenta di uno o più ordini di grandezza rispetto ai livelli fisiologici. Ciò provoca una competizione con l’ospite per i nutrienti e porta a sintomi gastrointestinali come la diarrea.[65]
Microbiota intestinale e dieta
La dieta è ampiamente riconosciuta come il fattore più influente in grado di modellare la composizione e l’attività metabolica del microbiota intestinale.[66] Sia la qualità dei nutrienti che la loro distribuzione nel tempo esercitano forti pressioni selettive, influenzando la diversità microbica dalla prima infanzia all’età adulta. Gli effetti della dieta possono essere osservati a più livelli, che vanno dalla colonizzazione iniziale alla configurazione enterotipica a lungo termine e alla modulazione del viroma intestinale.[15][55][67]
Ruolo del latte materno
Sebbene considerato sterile, il latte materno ospita un ricco microbiota composto da oltre 700 specie, dominato da stafilococchi, streptococchi, bifidobatteri e batteri lattici.[68] Dunque, nei bambini allattati al seno rappresenta una fonte importante per la colonizzazione dell’intestino.[32] È stato suggerito che questa modalità di colonizzazione sia strettamente correlata allo stato di salute del neonato, in quanto potrebbe proteggerlo dalle infezioni e contribuire alla maturazione del sistema immunitario.[69] Il latte materno influenza il microbiota intestinale anche indirettamente, grazie alla presenza di oligosaccaridi con attività prebiotica che stimolano la crescita di gruppi batterici specifici quali stafilococchi e bifidobatteri.[70]
Modelli dietetici in popolazioni differenti
Uno studio ha confrontato il microbiota intestinale di bambini europei e africani, rispettivamente da Firenze e un villaggio rurale del Burkina Faso, di età compresa tra 1 e 6 anni. Lo studio ha evidenziato il ruolo decisivo della dieta rispetto ad altre variabili quali il clima, la geografia, l’igiene e i servizi sanitari.[53]
È stata riportata una relativa convergenza funzionale, senza differenze significative nei principali percorsi immunologici, tra i due gruppi. Infatti, come ampiamente riportato in letteratura, i neonati, purché allattati al seno, presentano un microbiota intestinale molto simile, ricco di Actinobacteria, principalmente Bifidobacterium.[71]
La successiva introduzione di cibi solidi, ossia una dieta di tipo occidentale negli europei ricca in grassi e proteine animali, e una dieta povera in proteine animali ma ricca in carboidrati complessi nei bambini africani, porta a una differenziazione del rapporto Firmicutes/Bacteroidetes tra i due gruppi. Nei bambini europei erano più abbondanti i Gram-positivi, principalmente Firmicutes, mentre nei bambini africani prevalevano i Gram-negativi, principalmente Bacteroidetes.[72]
Effetti della dieta a lungo e breve termine
Modelli dietetici protratti a lungo termine sono fortemente associati alla ripartizione in enterotipi. Nello specifico:
- una dieta ricca in grassi e proteine animali, dunque di tipo occidentale, porta a un microbiota intestinale dominato da taxa dell’enterotipo Bacteroides;
- una dieta ricca in carboidrati, tipica delle società agricole, porta alla prevalenza dell’enterotipo Prevotella.[67]
Risultati analoghi sono emersi dallo studio sopracitato sui bambini. Negli europei, il microbiota intestinale era dominato da taxa tipici dell’enterotipo Bacteroides, mentre nei bambini del Burkina Faso dominavano taxa dell’enterotipo Prevotella.[72]
Cambiamenti a breve termine della dieta, circa 10 giorni, in particolare il passaggio da una povera in grassi e ricca in fibre a una ricca in grassi e povera in fibre e viceversa, provocano rapide modifiche nella composizione del microbioma, già dopo 24 ore. Tuttavia, questi cambiamenti non portano a modifiche stabili nella ripartizione dell’enterotipo, indicando che sono necessari modelli dietetici mantenuti nel lungo termine per indurre transizioni dell’enterotipo che permangano nel tempo.[73]
Dieta e viroma
Modifiche a carico della dieta si traducono anche in cambiamenti a carico del viroma intestinale. Questi cambiamenti comportano modifiche nelle proporzioni relative delle popolazioni virali preesistenti, verso le quali tendono a convergere individui che seguono la stessa dieta.[15][16]
Microbiota intestinale, stato di salute e malattie
Lo stato di salute sia del neonato che della madre nel corso della gravidanza rappresenta un fattore determinante della composizione del microbiota intestinale.[50] Ad esempio, in madri affette da malattia infiammatoria intestinale (IBD), una condizione caratterizzata da infiammazione intestinale cronica, il microbiota intestinale mostra una marcata deplezione di Faecalibacterium prausnitzii, una tra le specie produttrici di acido butirrico, un’importante fonte di energia per le cellule intestinali e con proprietà antinfiammatorie ben note. Questa riduzione è accompagnata da un aumento dell’abbondanza di Escherichia coli aderente, una firma microbica comunemente associata alla disbiosi intestinale.[74][75]
Microbiota intestinale, batteriocine e batteriofagi
Le batteriocine sono proteine dotate di attività antibatterica, mentre i batteriofagi svolgono un ruolo importante nel controllo dell’abbondanza e composizione del microbiota intestinale. In particolare, i batteriofagi potrebbero avere un ruolo di primo piano nel corso della colonizzazione dell’intestino del neonato infettando via via i batteri dominanti, creando così l’opportunità per un altro ceppo batterico di divenire prevalente.[76]
Questo modello di dinamiche preda–predatore, definito kill-the-winner model, suggerisce che le fioriture di determinate specie batteriche porterebbero a fioriture dei loro corrispondenti batteriofagi, seguite da un declino dell’abbondanza batterica. Di conseguenza, il genotipo fagico più abbondante cambia nel tempo. Sebbene alcune sequenze geniche nel viroma intestinale del neonato rimangano stabili durante i primi tre mesi di vita, sono state osservate drammatiche variazioni nella composizione complessiva della comunità virale tra la prima e la seconda settimana di vita. Nel corso di questo periodo anche la comunità batterica è estremamente dinamica.[17][37][77]
Microbiota intestinale e posizione geografica
È stato dimostrato che la posizione geografica influenza la composizione del microbiota intestinale fin dalla prima infanzia. È stata ad esempio osservata una sorta di gradiente geografico nel microbiota dei neonati europei, con un numero più elevato di specie del genere Bifidobacterium e alcune di Clostridium nei bambini delle zone settentrionali, mentre in quelli delle zone meridionali è stata rilevata una maggiore abbondanza di Bacteroides, Lactobacillus ed Eubacterium.[6][78]
Più in generale, studi condotti su popolazioni diverse indicano che la localizzazione geografica, insieme a fattori legati allo stile di vita quali dieta, modalità di sussistenza ed esposizioni ambientali, contribuisce in modo sostanziale alla variabilità nella composizione e nella diversità del microbiota intestinale tra le popolazioni.[79] Questi risultati suggeriscono che sia le influenze ambientali regionali sia le abitudini alimentari determinate dal contesto culturale svolgano un ruolo chiave nel modellare il microbioma intestinale nelle prime fasi di vita.
Bibliografia
- ^ Afzaal M., Saeed F., Shah Y.A., et al. Human gut microbiota in health and disease: unveiling the relationship. Front Microbiol 2022;13:999001. doi:10.3389/fmicb.2022.999001
- ^ Dekaboruah E., Suryavanshi M.V., Chettri D., Verma A.K. Human microbiome: an academic update on human body site specific surveillance and its possible role. Arch Microbiol 2020;202(8):2147-2167. doi:10.1007/s00203-020-01931-x
- ^ a b Ley R.E., Peterson D.A., and Gordon J.I. Ecological and evolutionary forces shaping microbial diversity in the human intestine. Cell 2006;124(4):837-848. doi:10.1016/j.cell.2006.02.017
- ^ Hitch T.C.A., Hall L.J., Walsh S.K., Leventhal G.E., Slack E., de Wouters T., Walter J., Clavel T. Microbiome-based interventions to modulate gut ecology and the immune system. Mucosal Immunol 2022;15(6):1095-1113. doi:10.1038/s41385-022-00564-1
- ^ a b c d Palmer C., Bik E.M., DiGiulio D.B., Relman D.A., and Brown P.O. Development of the human infant intestinal microbiota. PLoS Biol 2007;5(7):e177. doi:10.1371/journal.pbio.0050177
- ^ a b c d e Rodríguez J.M., Murphy K., Stanton C., et al. The composition of the gut microbiota throughout life, with an emphasis on early life. Microb Ecol Health Dis 2015;26:26050. doi:10.3402/mehd.v26.26050
- ^ a b c d Schoultz I., Claesson M.J., Dominguez-Bello M.G., et al. Gut microbiota development across the lifespan: disease links and health-promoting interventions. J Intern Med 2025;297(6):560-583. doi:10.1111/joim.20089
- ^ a b Maurer J.M., Schellekens R.C., van Rieke H.M., et al. Gastrointestinal pH and transit time profiling in healthy volunteers using the IntelliCap system confirms ileo-colonic release of ColoPulse tablets. PLoS One 2015;10(7):e0129076. doi:10.1371/journal.pone.0129076
- ^ Zhang J., Huang Y.J., Yoon J.Y., et al. Primary human colonic mucosal barrier crosstalk with super oxygen-sensitive Faecalibacterium prausnitzii in continuous culture. Med 2021;2(1):74-98.e9. doi:10.1016/j.medj.2020.07.001
- ^ a b Ma H., Wang K., Jiang C. Microbiota-derived bile acid metabolic enzymes and their impacts on host health. Cell Insight 2025;4(5):100265. doi:10.1016/j.cellin.2025.100265
- ^ Zeng Q., Feng X., Hu Y., Su S. The human gut microbiota is associated with host lifestyle: a comprehensive narrative review. Front Microbiol 2025;16:1549160. doi:10.3389/fmicb.2025.1549160
- ^ Qin J., Li R., Raes J., et al. A human gut microbial gene catalogue established by metagenomic sequencing. Nature 2010;464(7285):59-65. doi:10.1038/nature08821
- ^ Thursby E., Juge N. Introduction to the human gut microbiota. Biochem J 2017;474(11):1823-1836. doi:10.1042/BCJ20160510
- ^ Almeida A., Nayfach S., Boland M., et al. A unified catalog of 204,938 reference genomes from the human gut microbiome. Nat Biotechnol 2021;39(1):105-114. doi:10.1038/s41587-020-0603-3
- ^ a b c d Minot S., Sinha R., Chen J., Li H., Keilbaugh S.A., Wu G.D., Lewis J.D., and Bushman F.D. The human gut virome: inter-individual variation and dynamic response to diet. Genome Res 2011;21:1616-1625. doi:10.1101/gr.122705.111
- ^ a b Cao Z., Sugimura N., Burgermeister E., Ebert M.P., Zuo T., Lan P. The gut virome: a new microbiome component in health and disease. EBioMedicine 2022;81:104113. doi:10.1016/j.ebiom.2022.104113
- ^ a b c d Lim E.S., Zhou Y., Zhao G., Bauer I.K., et al. Early life dynamics of the human gut virome and bacterial microbiome in infants. Nat Med 2015;21(10):1228-34. doi:10.1038/nm.3950
- ^ Batinovic S., Wassef F., Knowler S.A., et al. Bacteriophages in natural and artificial environments. Pathogens 2019;8(3):100. doi:10.3390/pathogens8030100
- ^ Morgan X.C., Segata N., Huttenhower C. Biodiversity and functional genomics in the human microbiome. Trends Genet 2013;29(1):51-8. doi:10.1016/j.tig.2012.09.005
- ^ Arumugam M., Raes J., Pelletier E., et al. Enterotypes of the human gut microbiome. Nature 2011;473(7346):174-80. doi:10.1038/nature09944
- ^ Costea P.I., Hildebrand F., Arumugam M., et al. Enterotypes in the landscape of gut microbial community composition. Nat Microbiol 2018;3(1):8-16. doi:10.1038/s41564-017-0072-8
- ^ Francino M.P. Early development of the gut microbiota and immune health. Pathogens 2014;3(3):769-90. doi:10.3390/pathogens3030769
- ^ Gil A., Rueda R., Ozanne S.E., et al. Is there evidence for bacterial transfer via the placenta and any role in the colonization of the infant gut? – a systematic review. Crit Rev Microbiol 2020;46(5):493-507. doi:10.1080/1040841X.2020.1800587
- ^ Moles L., Gómez M., Heilig H., et al. Bacterial diversity in meconium of preterm neonates and evolution of their fecal microbiota during the first month of life. PLoS One 2013;8(6):e66986. doi:10.1371/journal.pone.0066986
- ^ Dominguez-Bello M.G., Costello E.K., Contreras M., Magris M., Hidalgo G., Fierer N., and Knight R. Delivery mode shapes the acquisition and structure of the initial microbiota across multiple body habitats in newborns. Proc Natl Acad Sci 2010;107:11971-11975. doi:10.1073/pnas.1002601107
- ^ Browne H.P., Neville B.A., Forster S.C., Lawley T.D. Transmission of the gut microbiota: spreading of health. Nat Rev Microbiol. 2017;15(9):531-543. doi:10.1038/nrmicro.2017.50
- ^ Liu T., Kress A.M., Debelius J., et al. Maternal vaginal and fecal microbiota in later pregnancy contribute to child fecal microbiota development in the ECHO cohort. iScience 2025;28(4):112211. doi:10.1016/j.isci.2025.112211
- ^ Rosenberg E., Zilber-Rosenberg I. Reconstitution and transmission of gut microbiomes and their genes between generations. Microorganisms 2021;10(1):70. doi:10.3390/microorganisms10010070
- ^ Goedert J.J. Intestinal microbiota and health of adults who were born by cesarean delivery. JAMA Pediatr 2016;170(10):1027. doi:10.1001/jamapediatrics.2016.2310
- ^ a b Zhang C., Li L., Jin B., Xu X., Zuo X., Li Y., Li Z. The effects of delivery mode on the gut microbiota and health: state of art. Front Microbiol 2021;12:724449. doi:10.3389/fmicb.2021.724449
- ^ Asnicar F., Manara S., Zolfo M., et al. Studying vertical microbiome transmission from mothers to infants by strain-level metagenomic profiling. mSystems 2017;2(1):e00164-16. doi:10.1128/mSystems.00164-16
- ^ a b Pannaraj P.S., Li F., Cerini C., et al. Association between breast milk bacterial communities and establishment and development of the infant gut microbiome. JAMA Pediatr 2017;171(7):647-654. doi:10.1001/jamapediatrics.2017.0378
- ^ Donald K., Finlay B.B. Early-life interactions between the microbiota and immune system: impact on immune system development and atopic disease. Nat Rev Immunol 2023;23(11):735-748. doi:10.1038/s41577-023-00874-w
- ^ Huurre A., Kalliomäki M., Rautava S., Rinne M., Salminen S., Isolauri E. Mode of delivery-effects on gut microbiota and humoral immunity. Neonatology 2008;93:236-240. doi:10.1159/000111102
- ^ Lai C., Huang L., Wang Y., Huang C., Luo Y., Qin X., Zeng J. Effect of different delivery modes on intestinal microbiota and immune function of neonates. Sci Rep 2024;14(1):17452. doi:10.1038/s41598-024-68599-x
- ^ Breitbart M., Haynes M., Kelley S., et al. Viral diversity and dynamics in an infant gut. Res Microbiol 2008;159:367-373. doi:10.1016/j.resmic.2008.04.006
- ^ a b Mills S., Shanahan F., Stanton C., Hill C., Coffey A., Ross R.P. Movers and shakers: influence of bacteriophages in shaping the mammalian gut microbiota. Gut Microbes 2013;4(1):4-16. doi:10.4161/gmic.22371
- ^ Redgwell T.A., Thorsen J., Petit M.A., et al. Prophages in the infant gut are pervasively induced and may modulate the functionality of their hosts. NPJ Biofilms Microbiomes 2025;11(1):46. doi:10.1038/s41522-025-00674-1
- ^ Koenig J.E., Spor A., Scalfone N., Fricker A.D., Stombaugh J., Knight R., Angenent L.T., and Ley R.E. Succession of microbial consortia in the developing infant gut microbiome. Proc Natl Acad Sci 2011;108(1):4578-4585. doi:10.1073/pnas.1000081107
- ^ Tarracchini C., Milani C., Lugli G.A., Mancabelli L., Turroni F., van Sinderen D., Ventura M. The infant gut microbiota as the cornerstone for future gastrointestinal health. Adv Appl Microbiol 2024;126:93-119. doi:10.1016/bs.aambs.2024.02.001
- ^ Saturio S., Nogacka A.M., Alvarado-Jasso G.M., Salazar N., de Los Reyes-Gavilán C.G., Gueimonde M., Arboleya S. Role of Bifidobacteria on infant health. Microorganisms 2021;9(12):2415. doi:10.3390/microorganisms9122415
- ^ Dargenio V.N., Cristofori F., Brindicci V.F., Schettini F., Dargenio C., Castellaneta S.P., Iannone A., Francavilla R. Impact of Bifidobacterium longum Subspecies infantis on pediatric gut health and nutrition: current evidence and future directions. Nutrients 2024;16(20):3510. doi:10.3390/nu16203510
- ^ Stinson L.F., Norrish I., Mhembere F., Cheema A.S., Mullally C.A., Payne M.S., Geddes D.T. Seeding and feeding: nutrition and birth-associated exposures shape gut microbiome assembly in breastfed infants. Gut Microbes 2025;17(1):2557981. doi:10.1080/19490976.2025.2557981
- ^ a b Kennedy E.A., Holtz L.R. Gut virome in early life: origins and implications. Curr Opin Virol 2022;55:101233. doi:10.1016/j.coviro.2022.101233
- ^ Shkoporov A.N., Clooney A.G., Sutton T.D.S., et al. The human gut virome is highly diverse, stable, and individual specific. Cell Host Microbe 2019;26(4):527-541.e5. doi:10.1016/j.chom.2019.09.009
- ^ Sekirov I., Russell S.L., Antunes L.C., Finlay B.B. Gut microbiota in health and disease. Physiol Rev 2010;90(3):859-904. doi:10.1152/physrev.00045.2009
- ^ Vallès Y., Artacho A., Pascual-García A., Ferrús M.L., Gosalbes M.J., Abellán J.J., Francino M.P. Microbial succession in the gut: directional trends of taxonomic and functional change in a birth cohort of Spanish infants. PLoS Genet 2014;10(6):e1004406. doi:10.1371/journal.pgen.1004406
- ^ Fusco W., Lorenzo M.B., Cintoni M., et al. Short-chain fatty-acid-producing bacteria: key components of the human gut microbiota. Nutrients 2023;15(9):2211. doi:10.3390/nu15092211
- ^ Derrien M., Alvarez A.S., de Vos W.M. The gut microbiota in the first decade of life. Trends Microbiol 2019;27(12):997-1010. doi:10.1016/j.tim.2019.08.001
- ^ a b Yao Y., Cai X., Ye Y., Wang F., Chen F., Zheng C. The role of microbiota in infant health: from early life to adulthood. Front Immunol 2021;12:708472. doi:10.3389/fimmu.2021.708472
- ^ a b Hou K., Wu Z.X., Chen X.Y., et al. Microbiota in health and diseases. Signal Transduct Target Ther 2022;7(1):135. doi:10.1038/s41392-022-00974-4
- ^ Augustynowicz G., Lasocka M., Szyller H.P., Dziedziak M., Mytych A., Braksator J., Pytrus T. The role of gut microbiota in the development and treatment of obesity and overweight: a literature review. J Clin Med 2025;14(14):4933. doi:10.3390/jcm14144933
- ^ a b De Filippo C., Cavalieri D., Di Paola M., et al. Impact of diet in shaping gut microbiota revealed by a comparative study in children from Europe and rural Africa. Proc Natl Acad Sci 2010;107(33):14691-14696. doi:10.1073/pnas.1005963107
- ^ Sharon I., Quijada N.M., Pasolli E., et al. The core human microbiome: does it exist and how can we find it? A critical review of the concept. Nutrients 2022;14(14):2872. doi:10.3390/nu14142872
- ^ a b David L.A., Maurice C.F., Carmody R.N., et al. Diet rapidly and reproducibly alters the human gut microbiome. Nature 2014;505(7484):559-63. doi:10.1038/nature12820
- ^ Sommer F., Anderson J.M., Bharti R., Raes J., Rosenstiel P. The resilience of the intestinal microbiota influences health and disease. Nat Rev Microbiol 2017;15(10):630-638. doi:10.1038/nrmicro.2017.58
- ^ Mangiola F., Nicoletti A., Gasbarrini A., Ponziani F.R. Gut microbiota and aging. Eur Rev Med Pharmacol Sci 2018;22(21):7404-7413. doi:10.26355/eurrev_201811_16280
- ^ Claesson M.J., Cusack S., O’Sullivan O., et al. Composition, variability, and temporal stability of the intestinal microbiota of the elderly. Proc Natl Acad Sci USA 2011;108(Suppl 1);4586-4591. doi:10.1073/pnas.1000097107
- ^ Salazar N., Valdés-Varela L., González S., Gueimonde M., de Los Reyes-Gavilán C.G. Nutrition and the gut microbiome in the elderly. Gut Microbes. 2017;8(2):82-97. doi:10.1080/19490976.2016.1256525
- ^ Santacroce L., Topi S., Bottalico L., Charitos I.A., Jirillo E. Current knowledge about gastric microbiota with special emphasis on Helicobacter pylori-related gastric conditions. Curr Issues Mol Biol 2024;46(5):4991-5009. doi:10.3390/cimb46050299
- ^ Yang K., Li G., Li Q., et al. Distribution of gut microbiota across intestinal segments and their impact on human physiological and pathological processes. Cell Biosci 2025;15(1):47. doi:10.1186/s13578-025-01385-y
- ^ Portincasa P., Bonfrate L., Khalil M., Angelis M., Calabrese F.M., D’Amato M., Wang D.Q., Di Ciaula A. Intestinal barrier and permeability in health, obesity and NAFLD. Biomedicines 2021;10(1):83. doi:10.3390/biomedicines10010083
- ^ Kastl A.J. Jr, Terry N.A., Wu G.D., Albenberg L.G. The structure and function of the human small intestinal microbiota: current understanding and future directions. Cell Mol Gastroenterol Hepatol 2020;9(1):33-45. doi:10.1016/j.jcmgh.2019.07.006
- ^ Donaldson G.P., Lee S.M., Mazmanian S.K. Gut biogeography of the bacterial microbiota. Nat Rev Microbiol 2016;14(1):20-32. doi:10.1038/nrmicro3552
- ^ Leite G., Morales W., Weitsman S., Celly S, et al. The duodenal microbiome is altered in small intestinal bacterial overgrowth. PLoS One 2020;15(7):e0234906. doi:10.1371/journal.pone.0234906
- ^ Zhang L., Tuoliken H., Li J., Gao H. Diet, gut microbiota, and health: a review. Food Sci Biotechnol 2024;34(10):2087-2099. doi:10.1007/s10068-024-01759-x
- ^ a b Wu G.D., Chen J., Hoffmann C., et al. Linking long-term dietary patterns with gut microbial enterotypes. Science 2011;334:105-108. doi:10.1126/science.1208344
- ^ Consales A., Cerasani J., Sorrentino G., Morniroli D., Colombo L., Mosca F., Giannì M.L. The hidden universe of human milk microbiome: origin, composition, determinants, role, and future perspectives. Eur J Pediatr 2022;181(5):1811-1820. doi:10.1007/s00431-022-04383-1
- ^ Fernández L., Langa S., Martín V., Maldonado A., Jiménez E., Martín R., Rodríguez J.M. The human milk microbiota: origin and potential roles in health and disease. Pharmacol Res 2013;69(1):1-10. doi:10.1016/j.phrs.2012.09.001
- ^ Newburg D.S., Morelli L. Human milk and infant intestinal mucosal glycans guide succession of the neonatal intestinal microbiota. Pediatr Res 2015;77:115-120. doi:10.1038/pr.2014.178
- ^ Donald K., Finlay B.B. Early-life interactions between the microbiota and immune system: impact on immune system development and atopic disease. Nat Rev Immunol 2023;23(11):735-748. doi:10.1038/s41577-023-00874-w
- ^ a b De Filippo C., Di Paola M., Ramazzotti M., et al. Diet, environments, and gut microbiota. A preliminary investigation in children living in rural and urban Burkina Faso and Italy. Front Microbiol 2017;8:1979. doi:10.3389/fmicb.2017.01979
- ^ Rodriguez C.I., Isobe K., Martiny J.B.H. Short-term dietary fiber interventions produce consistent gut microbiome responses across studies. mSystems 2024;9(6):e0013324. doi:10.1128/msystems.00133-24
- ^ Cao Y., Shen J., Ran Z.H. Association between Faecalibacterium prausnitzii reduction and inflammatory bowel disease: a meta-analysis and systematic review of the literature. Gastroenterol Res Pract 2014;2014:872725. doi:10.1155/2014/872725
- ^ Lavelle A., Sokol H. Gut microbiota-derived metabolites as key actors in inflammatory bowel disease. Nat Rev Gastroenterol Hepatol 2020;17(4):223-237. doi:10.1038/s41575-019-0258-z
- ^ Emencheta S.C., Olovo C.V., Eze O.C., et al. The role of bacteriophages in the gut microbiota: implications for human health. Pharmaceutics 2023;15(10):2416. doi:10.3390/pharmaceutics15102416
- ^ Mills S., Ross R.P., Hill C. Bacteriocins and bacteriophage; a narrow-minded approach to food and gut microbiology. FEMS Microbiol Rev 2017;41(1):S129-S153. doi:10.1093/femsre/fux022
- ^ Fallani M., Young D., Scott J., et al. Intestinal microbiota of 6-week-old infants across Europe: geographic influence beyond delivery mode, breast-feeding, and antibiotics. J Pediatr Gastroenterol Nutr 2010;51(1):77-84. doi:10.1097/MPG.0b013e3181d1b11e
- ^ Gupta V.K., Paul S., Dutta C. Geography, ethnicity or subsistence-specific variations in human microbiome composition and diversity. Front Microbiol 2017;8:1162. doi:10.3389/fmicb.2017.01162